julio 28, 2021
~ 9 MIN
Pytorch + Docker
< Blog RSSPytorch + Docker
En los posts anteriores hemos visto muchos trucos para optimizar nuestro código en Pytorch, sin embargo no nos hemos preocupado por la instalación del mismo. En este post vamos a aprender a usar una versión de Pytorch optimizada que en alguna ocasión nos dará un pequeño extra de performance. Para ello vamos a usar Docker.
¿Qué es Docker?
Docker es una tecnología que nos permite crear entornos de software paquetizados y aisaldos del resto de componenetes en un sistema operativo. Similar en espíritu a una máquina virtual, o a los entornos virtuales de Python, con Docker podremos crear imágenes (que contendrán nuestro código y sus dependencias) que serán ejectuadas en contenedores (el entorno aislado). De esta manera estremos seguros que nuestro código funcionará en cualquier plataforma siempre y cuando Docker esté instalado.
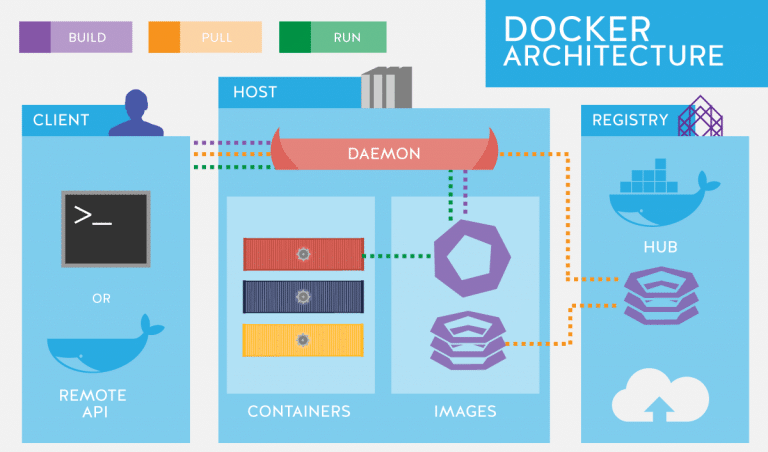
No es el objetivo de este post explicar en detalle cómo trabajar con Docker, para ello existen multitud de tutoriales en internet. Pero sí que vamos a ver como podemos aplicar esta tecnlogía a la hora de trabajar con Pytorch.
⚠️ Si bien puedes instalar
Dockeren cualquier sistema operativo, lo que vamos a ver en este post sólo funciona en Linux.
Dependencias
Para poder trabajar con Pytorch y Docker necesitamos instalar las siguientes dependencias:
Dockernvidia-docker(para queDockerpuedas usar nuestraGPU)- Opcionalmente, instala
docker-compose
Ejecutando Jupyter Notebooks en Docker
Lo primero que vamos a ver es como ejecutar una imágen de Docker con Python. Para ello, una vez hayas instalado Docker, ejecuta el siguiente comando en la terminal:
docker run -it python:3.9-slim python
Esto descargará la imágen python:3.9-slim de dockerhub si no la encuentra en el sistema y luego ejecutará el comando python. Al haber indicado a Docker que lo ejecute en modo interactivo (-it) se abrirá un terminal de Python dentro del contenedor. Como puedes ver, si necesitas una versión de Python diferente simplemente tendrás que indicarle el tag apropiado.
Para abrir un notebook puedes usar el comando siguiente:
docker run python:3.9-slim jupyter notebook
sin embargo esto dará un error, y es que la imágen usada no tiene jupyter notebook instalado. Tenemos dos opciones, usar una imágen diferente que sí lo tenga o bien crearnos nuestra propia imágen. Para ello, crea un archivo llamado Dockerfile con el siguiente contenido:
FROM python:3.9-slim
RUN pip install jupyter
WORKDIR /workspace
Ahora puedes crear tu propia imágen con el comando
docker build -t jupyter .
Y ejectuarla de la siguiente manera
docker run jupyter jupyter notebook --allow-root
Sin embargo, esto seguirá sin funcionar. El motivo es que nuestra imágen está siendo ejectuada en un entorno aisaldo, en el que no existe un navegador ni tampoco forma de poder conectarnos. Para solventar este problema ejecutaremos el comando
docker run -p 8888:8888 jupyter jupyter notebook --allow-root --no-browser --ip=0.0.0.0
Ahora ya podrás usar tus notebooks !!! Sin embargo, si creas un nuevo notebook y paras el contenedor, al volver a abrirlo habrá desaparecido. Esto es debido a que, como ya hemos comentado, nuestro código se ejecuta en un entorno aislado por lo que no tendremos persistencia en nuestros archivos. Para solventarlo, usarmos el conceptro de volumes (carpetas compartidas entre nuestro ordenador y Docker).
docker run -p 8888:8888 -v ${PWD}/ipynbs:/workspace/ipynbs jupyter jupyter notebook --allow-root --no-browser --ip=0.0.0.0
Con esto tenemos todo lo necesario para trabajar con notebooks en Docker. Te recomiendo como último paso utilizas docker-compose ya que tener que ejecutar un comando tan largo en la consola puede convertirse en algo tedioso. Docker-compose es una herramienta que nos permite ejecutar nuestros contenedores usando archivos de configuración (entre muchas otras cosas que no veremos aquí). Para ello, crea un archivo llamado docker-compose.yml con el siguiente contenido:
version: "3.0"
services:
jupyter:
container_name: jupyter
image: jupyter
build: .
ports:
- 8888:8888
volumes:
- ./ipynbs:/workspace/ipynbs
command: jupyter notebook --allow-root --ip=0.0.0.0 --no-browser
Puedes crear tus imágenes con el comando docker-compose build, ejecutar el contenedor con docker-compose up y pararlo con docker-compose down.
Pytorch en Docker
Vamos ahora a ver cómo podemos trabajar con Pytorch en Docker. Si bien podemos hacer como antes e instalar Pytorch en nuestra imágen, ahora haremos lo contrario y usaremos una imágen ya hecha. El motivo es que podemos encontrar imágenes en internet con instalaciones optimizadas que nos pueden dar un extra de performance en ciertos casos gracias al acceso a nuevas características o funcionalidad no implementadas en la distribución oficial que podemos instalar con conda. Un repositorio muy recomendado en ngc, en el que podemos encontrar instalaciones de Pytorch opimizadas por los propios ingenieros de NVIDIA. Puedes ejecutar un notebook con esta imágen con el siguiente docker-compose.
version: "2.3"
services:
pytorch-ngc:
runtime: nvidia
container_name: pytorch-ngc
image: nvcr.io/nvidia/pytorch:21.06-py3
ipc: host
ports:
- 8888:8888
volumes:
- ./ipynbs:/workspace
- ./data:/workspace/data
environment:
- NVIDIA_VISIBLE_DEVICES=all
command: jupyter notebook --allow-root --ip=0.0.0.0 --no-browser --NotebookApp.token=abc123
Aunque es muy probable que en algún momento quieras customizar esta imágen para incluir alguna librería extra, como por ejemplo Pytorch Lightning (si bien existe una imágen con esta librería instalada, vamos a ver un ejemplo de cómo puedes añadir tus propias dependencias). Para ello, ya sabes que tienes que crear un Dockerfile.
FROM nvcr.io/nvidia/pytorch:21.06-py3
RUN pip install --upgrade pip && pip install \
pytorch-lightning \
timm \
pytorch-segmentation-models
WORKDIR /workspace
Y luegos puedes usar el siguiente docker-compose.
version: "2.3"
services:
pytorch-ngc:
runtime: nvidia
container_name: pytorch-ngc
image: pytorch-ngc
build: .
ipc: host
ports:
- 8888:8888
volumes:
- ./ipynbs:/workspace
- ./data:/workspace/data
environment:
- NVIDIA_VISIBLE_DEVICES=all
command: jupyter notebook --allow-root --ip=0.0.0.0 --no-browser --NotebookApp.token=abc123
Una vez hayas jugado un poco con todo lo visto hasta ahora, puedes intentar abrir este mismo notebook con Docker y ejectuar el siguiente código para entrenar una red neuronal. Para ello necesitarás descargar el dataset EuroSAT y colocarlo en la carpeta data.
import torch
torch.__version__
'1.9.0'
torch.cuda.is_available()
True
import os
from sklearn.model_selection import train_test_split
def setup(path='./data', test_size=0.2, random_state=42):
classes = sorted(os.listdir(path))
print("Generating images and labels ...")
images, encoded = [], []
for ix, label in enumerate(classes):
_images = os.listdir(f'{path}/{label}')
images += [f'{path}/{label}/{img}' for img in _images]
encoded += [ix]*len(_images)
print(f'Number of images: {len(images)}')
# train / val split
print("Generating train / val splits ...")
train_images, val_images, train_labels, val_labels = train_test_split(
images,
encoded,
stratify=encoded,
test_size=test_size,
random_state=random_state
)
print("Training samples: ", len(train_labels))
print("Validation samples: ", len(val_labels))
return classes, train_images, train_labels, val_images, val_labels
classes, train_images, train_labels, val_images, val_labels = setup('./data')
Generating images and labels ...
Number of images: 27000
Generating train / val splits ...
Training samples: 21600
Validation samples: 5400
import torch
from skimage import io
class Dataset(torch.utils.data.Dataset):
def __init__(self, images, labels):
self.images = images
self.labels = labels
def __len__(self):
return len(self.images)
def __getitem__(self, ix):
img = io.imread(self.images[ix])[...,(3,2,1)]
img = torch.tensor(img / 4000, dtype=torch.float).clip(0,1).permute(2,0,1)
label = torch.tensor(self.labels[ix], dtype=torch.long)
return img, label
ds = {
'train': Dataset(train_images, train_labels),
'val': Dataset(val_images, val_labels)
}
batch_size = 1024
dl = {
'train': torch.utils.data.DataLoader(ds['train'], batch_size=batch_size, shuffle=True, num_workers=20, pin_memory=True),
'val': torch.utils.data.DataLoader(ds['val'], batch_size=batch_size, shuffle=False, num_workers=20, pin_memory=True)
}
import torch.nn.functional as F
import timm
class Model(torch.nn.Module):
def __init__(self, n_outputs=10, use_amp=True):
super().__init__()
self.model = timm.create_model('tf_efficientnet_b5', pretrained=True, num_classes=n_outputs)
self.use_amp = use_amp
def forward(self, x, log=False):
if log:
print(x.shape)
with torch.cuda.amp.autocast(enabled=self.use_amp):
return self.model(x)
from tqdm import tqdm
import numpy as np
def step(model, batch, device):
x, y = batch
x, y = x.to(device), y.to(device)
y_hat = model(x)
loss = F.cross_entropy(y_hat, y)
acc = (torch.argmax(y_hat, axis=1) == y).sum().item() / y.size(0)
return loss, acc
def train_amp(model, dl, optimizer, epochs=10, device="cpu", use_amp = True, prof=None, end=0):
model.to(device)
hist = {'loss': [], 'acc': [], 'val_loss': [], 'val_acc': []}
scaler = torch.cuda.amp.GradScaler(enabled=use_amp)
for e in range(1, epochs+1):
# train
model.train()
l, a = [], []
bar = tqdm(dl['train'])
stop=False
for batch_idx, batch in enumerate(bar):
optimizer.zero_grad()
# AMP
with torch.cuda.amp.autocast(enabled=use_amp):
loss, acc = step(model, batch, device)
scaler.scale(loss).backward()
# gradient clipping
#torch.nn.utils.clip_grad_norm_(model.parameters(), max_norm=0.1)
scaler.step(optimizer)
scaler.update()
l.append(loss.item())
a.append(acc)
bar.set_description(f"training... loss {np.mean(l):.4f} acc {np.mean(a):.4f}")
# profiling
if prof:
if batch_idx >= end:
stop = True
break
prof.step()
hist['loss'].append(np.mean(l))
hist['acc'].append(np.mean(a))
if stop:
break
# eval
model.eval()
l, a = [], []
bar = tqdm(dl['val'])
with torch.no_grad():
for batch in bar:
loss, acc = step(model, batch, device)
l.append(loss.item())
a.append(acc)
bar.set_description(f"evluating... loss {np.mean(l):.4f} acc {np.mean(a):.4f}")
hist['val_loss'].append(np.mean(l))
hist['val_acc'].append(np.mean(a))
# log
log = f'Epoch {e}/{epochs}'
for k, v in hist.items():
log += f' {k} {v[-1]:.4f}'
print(log)
return hist
model = Model()
optimizer = torch.optim.Adam(model.parameters(), lr=1e-3)
hist = train_amp(model, dl, optimizer, epochs=3, device="cuda")
training... loss 1.4168 acc 0.7123: 100%|██████████| 22/22 [00:11<00:00, 1.88it/s]
evluating... loss 7.0801 acc 0.3112: 100%|██████████| 6/6 [00:01<00:00, 3.25it/s]
0%| | 0/22 [00:00<?, ?it/s]
Epoch 1/3 loss 1.4168 acc 0.7123 val_loss 7.0801 val_acc 0.3112
training... loss 0.1586 acc 0.9484: 100%|██████████| 22/22 [00:10<00:00, 2.05it/s]
evluating... loss 0.4077 acc 0.8919: 100%|██████████| 6/6 [00:01<00:00, 3.26it/s]
0%| | 0/22 [00:00<?, ?it/s]
Epoch 2/3 loss 0.1586 acc 0.9484 val_loss 0.4077 val_acc 0.8919
training... loss 0.0436 acc 0.9868: 100%|██████████| 22/22 [00:10<00:00, 2.02it/s]
evluating... loss 0.2022 acc 0.9483: 100%|██████████| 6/6 [00:01<00:00, 3.28it/s]
Epoch 3/3 loss 0.0436 acc 0.9868 val_loss 0.2022 val_acc 0.9483
Scripts en Docker
Si bien trabajar con notebooks es muy conveniente en la mayoría de ocasiones, es posible que alternativamente quieras ejectuar un script dentro del contenedor de Docker. Puedes usar un volume para cargar tu código en el contenedor (ideal durante el desarrollo) o directamente copiar el script en la imágen (ideal si quieres compartir tu código o ejectuarlo en máquinas remotas).
FROM nvcr.io/nvidia/pytorch:21.06-py3
RUN pip install --upgrade pip && pip install \
pytorch-lightning \
timm \
segmentation-models-pytorch
COPY ./main.py /workspace/main.py
WORKDIR /workspace
Los datos los seguirás teniendo que cargar en un volume ya que meterlos en la imágen sería algo feo 😛, igualmente asegúrate de usar otro volume en el que guardar tus resultados.
version: "2.3"
services:
pytorch-script:
runtime: nvidia
container_name: pytorch-script
image: pytorch-script
build: .
ipc: host
volumes:
- ./data:/workspace/data
environment:
- NVIDIA_VISIBLE_DEVICES=all
command: python main.py
Resumen
Docker es una herramienta muy útil a la hora de gestionar nuestro código y sus dependencias. En este post hemos visto un ejemplo de cómo usarlo junto a Pytorch, pero lo puedes usar para muchas otras aplicaciones (webs, bases de datos, ...). Hemos aprendido a ejectuar notebooks dentro de contenedores (para lo cual necesitamos configurar los puertos y volúmenes adecuados), usar versiones optimizadas de Pytorch gracias al NGC de NVIDIA y crear nuestras propias imágenes con nuestro código y dependencias.