junio 6, 2022
~ 15 MIN
ML - Support Vector Machines
< Blog RSSML - Support Vector Machine
En posts anteriores hemos aprendido sobre las dos tareas más típicas en el mundo del Machine Learning: la regresión y la classificación, y un primer modelo sencillo para atacar estos problemas: la regresión lineal (o regresión logísitica en el caso de la clasificación). Si bien este modelo es muy sencillo, eficiente y explicable (cada variable tiene un peso asignado, por lo que podemos saber cuánto contribuye a la decisión final) su principal limitación se encuentra, como ya vimos, en que no es capaz de aprender datasets que no sigan tendencias lineales. Esto motivó el desarrollo de nuevos modelos de ML capaces de sobreponerse a estas limitaciones. Entre ellos, uno de los más usados por sus buenas prestaciones cuando no disponemos de muchos datos son las máquinas de soporte vectorial, o Support Vector Machines (SVM) en inglés.
Clasificación lineal con SVMs
Mientras que los modelos de regresión lineal se basan en la idea de minimizar el número de errores, las SVMs intentan maximizar la distancia entre las fronteras de decisión y los datos de entrenamiento: el margen.

Añadir más datos fuera del margen no afecta al modelo ya que está totalmente determinado por las instancias localizadas en los límites de la frontera: los vectores soporte. Esto solo funciona si los datos son linealmente separables, en caso contrario deberemos aceptar un cierto número de errores (aunque en la sección siguiente veremos como hacerlo con un modelo no lineal).
En Scikit-Learn puedes usar el modelo LinearSVC para entrenar un clasificador lineal con SVMs. En el siguiente ejemplo veremos como podemos ajustar el número de errores aceptables en la clasificación (instancias dentro del margen) con el dataset Iris, que ya hemos usado en varios de los posts anteriores.
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.svm import LinearSVC
# cargar dataset (usamos dos features y una sola clase)
iris = datasets.load_iris()
X = iris["data"][:, (2, 3)] # petal length, petal width
y = (iris["target"] == 2).astype(np.float64) # Iris virginica
# normalizar datos
scaler = StandardScaler()
# SVMs (dos diferentes valires de C)
svm_clf1 = LinearSVC(C=1, loss="hinge", random_state=42)
svm_clf2 = LinearSVC(C=100, loss="hinge", random_state=42)
# entrenar modelos
scaled_svm_clf1 = Pipeline([
("scaler", scaler),
("linear_svc", svm_clf1),
])
scaled_svm_clf2 = Pipeline([
("scaler", scaler),
("linear_svc", svm_clf2),
])
scaled_svm_clf1.fit(X, y)
scaled_svm_clf2.fit(X, y)
/home/juan/miniconda3/lib/python3.8/site-packages/sklearn/svm/_base.py:1206: ConvergenceWarning: Liblinear failed to converge, increase the number of iterations.
warnings.warn(
Pipeline(steps=[('scaler', StandardScaler()),
('linear_svc',
LinearSVC(C=100, loss='hinge', random_state=42))])
# extraer fronteras de decisión (desnormalizadas)
b1 = svm_clf1.decision_function([-scaler.mean_ / scaler.scale_])
b2 = svm_clf2.decision_function([-scaler.mean_ / scaler.scale_])
w1 = svm_clf1.coef_[0] / scaler.scale_
w2 = svm_clf2.coef_[0] / scaler.scale_
svm_clf1.intercept_ = np.array([b1])
svm_clf2.intercept_ = np.array([b2])
svm_clf1.coef_ = np.array([w1])
svm_clf2.coef_ = np.array([w2])
# Calcular vectores soporte
t = y * 2 - 1
support_vectors_idx1 = (t * (X.dot(w1) + b1) < 1).ravel()
support_vectors_idx2 = (t * (X.dot(w2) + b2) < 1).ravel()
svm_clf1.support_vectors_ = X[support_vectors_idx1]
svm_clf2.support_vectors_ = X[support_vectors_idx2]
# visualizar fronteras de decisión
def plot_svc_decision_boundary(svm_clf, xmin, xmax):
w = svm_clf.coef_[0]
b = svm_clf.intercept_[0]
x0 = np.linspace(xmin, xmax, 200)
decision_boundary = -w[0]/w[1] * x0 - b/w[1]
margin = 1/w[1]
gutter_up = decision_boundary + margin
gutter_down = decision_boundary - margin
svs = svm_clf.support_vectors_
plt.scatter(svs[:, 0], svs[:, 1], s=180, facecolors='#FFAAAA')
plt.plot(x0, decision_boundary, "k-", linewidth=2)
plt.plot(x0, gutter_up, "k--", linewidth=2)
plt.plot(x0, gutter_down, "k--", linewidth=2)
fig, axes = plt.subplots(ncols=2, figsize=(10,2.7), sharey=True)
plt.sca(axes[0])
plt.plot(X[:, 0][y==1], X[:, 1][y==1], "g^", label="Iris virginica")
plt.plot(X[:, 0][y==0], X[:, 1][y==0], "bs", label="Iris versicolor")
plot_svc_decision_boundary(svm_clf1, 4, 5.9)
plt.xlabel("Petal length", fontsize=14)
plt.ylabel("Petal width", fontsize=14)
plt.legend(loc="upper left", fontsize=14)
plt.title("$C = {}$".format(svm_clf1.C), fontsize=16)
plt.axis([4, 5.9, 0.8, 2.8])
plt.sca(axes[1])
plt.plot(X[:, 0][y==1], X[:, 1][y==1], "g^")
plt.plot(X[:, 0][y==0], X[:, 1][y==0], "bs")
plot_svc_decision_boundary(svm_clf2, 4, 5.99)
plt.xlabel("Petal length", fontsize=14)
plt.title("$C = {}$".format(svm_clf2.C), fontsize=16)
plt.axis([4, 5.9, 0.8, 2.8])
plt.show()
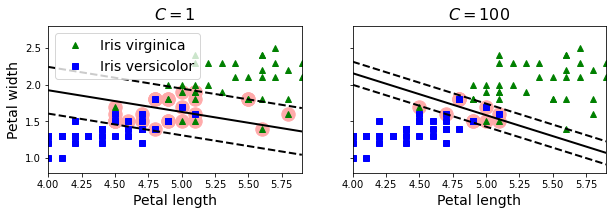
Para saber si una instancia pertenece a una clase u otra, podemos calcular la frontera de decición , donde son los parámteros del modelo, y es el bias. Luego, podemos asignar la clase en función de