junio 17, 2022
~ 14 MIN
ML - Aprendizaje no supervisado
< Blog RSSML - Aprendizaje No Supervisado
Si bien todos los ejemplos de Machine Learning que hemos visto hasta ahora en esta serie se han basado en aprendizaje supervisado (nuestros datos van acompañados de las etiquetas correspondientes, ejemplos de la tarea que queremos llevar a cabo) la mayoría de los datos no están etiquetados. Antes esta problemática tenemos dos alternativas: etiquetar datos manualmente (lo cual require de tiempo, esfuerzo y dinero en el caso de querer desarrollar sistemas reales) o bien usar técnicas de aprendizaje no supervisado, o Unsupervised Learning en inglés.
Clustering
De entre las diferentes técnicas de aprendizaje no supervisado, el Clustering es una de las más usadas. Este técnica consiste en identificar aquellas muestras similares del conjunto de datos y asignarlas a un cluster, o grupo. Esto permite aplicaciones tales como: segmentación de clientes, análisis de datos, reducción de la dimensionalidad, detección de anomalías, aprendizaje semi-supervisado, motores de búsqueda e incluso la semgentación de imágenes.
K-Means
El algoritmo de K-Means es uno de los más usados a la hora de aplicar Clustering, ya que es un método rápido y eficiente. Vamos a generar un conjunto de datos sintético para aprender a usar esta técnica.
from sklearn.datasets import make_blobs
import numpy as np
blob_centers = np.array(
[[ 0.2, 2.3],
[-1.5 , 2.3],
[-2.8, 1.8],
[-2.8, 2.8],
[-2.8, 1.3]])
blob_std = np.array([0.4, 0.3, 0.1, 0.1, 0.1])
X, y = make_blobs(n_samples=2000, centers=blob_centers,
cluster_std=blob_std, random_state=7)
import matplotlib.pyplot as plt
def plot_clusters(X, y=None):
plt.scatter(X[:, 0], X[:, 1], c=y, s=1)
plt.xlabel("$x_1$", fontsize=14)
plt.ylabel("$x_2$", fontsize=14, rotation=0)
plt.figure(figsize=(8, 4))
plot_clusters(X)
plt.show()
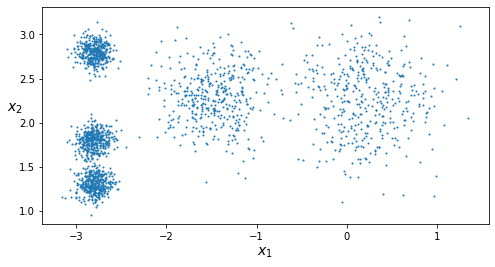
En Scikit-Learn puedes usar el objeto KMeans, indicando el número de clusters deseado.
from sklearn.cluster import KMeans
k = 5
kmeans = KMeans(n_clusters=k, random_state=42)
y_pred = kmeans.fit_predict(X)
y_pred
array([4, 0, 1, ..., 2, 1, 0], dtype=int32)
kmeans.cluster_centers_
array([[-2.80389616, 1.80117999],
[ 0.20876306, 2.25551336],
[-2.79290307, 2.79641063],
[-1.46679593, 2.28585348],
[-2.80037642, 1.30082566]])
X_new = np.array([[0, 2], [3, 2], [-3, 3], [-3, 2.5]])
kmeans.predict(X_new)
array([1, 1, 2, 2], dtype=int32)
def plot_data(X):
plt.plot(X[:, 0], X[:, 1], 'k.', markersize=2)
def plot_centroids(centroids, weights=None, circle_color='w', cross_color='k'):
if weights is not None:
centroids = centroids[weights > weights.max() / 10]
plt.scatter(centroids[:, 0], centroids[:, 1],
marker='o', s=10, linewidths=8,
color=circle_color, zorder=10, alpha=0.9)
plt.scatter(centroids[:, 0], centroids[:, 1],
marker='x', s=2, linewidths=10,
color=cross_color, zorder=11, alpha=1)
def plot_decision_boundaries(clusterer, X, resolution=1000, show_centroids=True,
show_xlabels=True, show_ylabels=True):
mins = X.min(axis=0) - 0.1
maxs = X.max(axis=0) + 0.1
xx, yy = np.meshgrid(np.linspace(mins[0], maxs[0], resolution),
np.linspace(mins[1], maxs[1], resolution))
Z = clusterer.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(Z, extent=(mins[0], maxs[0], mins[1], maxs[1]),
cmap="Pastel2")
plt.contour(Z, extent=(mins[0], maxs[0], mins[1], maxs[1]),
linewidths=1, colors='k')
plot_data(X)
if show_centroids:
plot_centroids(clusterer.cluster_centers_)
if show_xlabels:
plt.xlabel("$x_1$", fontsize=14)
else:
plt.tick_params(labelbottom=False)
if show_ylabels:
plt.ylabel("$x_2$", fontsize=14, rotation=0)
else:
plt.tick_params(labelleft=False)
plt.figure(figsize=(8, 4))
plot_decision_boundaries(kmeans, X)
plt.show()
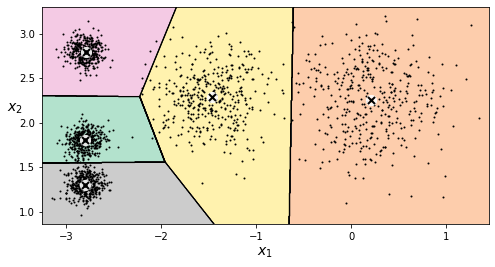
En lugar de asignar un grupo a cada muestra (hard clustering) se le puede asignar una puntuación (soft clustering) en función de su distancia al centroide.
kmeans.transform(X_new)
array([[2.81093633, 0.32995317, 2.9042344 , 1.49439034, 2.88633901],
[5.80730058, 2.80290755, 5.84739223, 4.4759332 , 5.84236351],
[1.21475352, 3.29399768, 0.29040966, 1.69136631, 1.71086031],
[0.72581411, 3.21806371, 0.36159148, 1.54808703, 1.21567622]])
Este algoritmo funciona asignando centroides de manera aleatoria y, de manera iterativa, asigna cada instancia a uno de ellos (el más cercano) tras lo cual se recalcula el centroide como el valor promedio de todas las instancias asignadas al grupo.
kmeans_iter1 = KMeans(n_clusters=5, init="random", n_init=1,
algorithm="full", max_iter=1, random_state=1)
kmeans_iter2 = KMeans(n_clusters=5, init="random", n_init=1,
algorithm="full", max_iter=2, random_state=1)
kmeans_iter3 = KMeans(n_clusters=5, init="random", n_init=1,
algorithm="full", max_iter=3, random_state=1)
kmeans_iter1.fit(X)
kmeans_iter2.fit(X)
kmeans_iter3.fit(X)
KMeans(algorithm='full', init='random', max_iter=3, n_clusters=5, n_init=1,
random_state=1)
plt.figure(figsize=(10, 8))
plt.subplot(321)
plot_data(X)
plot_centroids(kmeans_iter1.cluster_centers_, circle_color='r', cross_color='w')
plt.ylabel("$x_2$", fontsize=14, rotation=0)
plt.tick_params(labelbottom=False)
plt.title("Update the centroids (initially randomly)", fontsize=14)
plt.subplot(322)
plot_decision_boundaries(kmeans_iter1, X, show_xlabels=False, show_ylabels=False)
plt.title("Label the instances", fontsize=14)
plt.subplot(323)
plot_decision_boundaries(kmeans_iter1, X, show_centroids=False, show_xlabels=False)
plot_centroids(kmeans_iter2.cluster_centers_)
plt.subplot(324)
plot_decision_boundaries(kmeans_iter2, X, show_xlabels=False, show_ylabels=False)
plt.subplot(325)
plot_decision_boundaries(kmeans_iter2, X, show_centroids=False)
plot_centroids(kmeans_iter3.cluster_centers_)
plt.subplot(326)
plot_decision_boundaries(kmeans_iter3, X, show_ylabels=False)
plt.show()
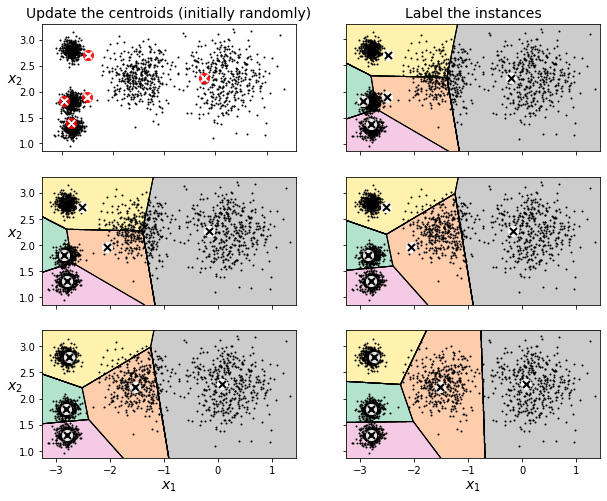
Esto implica que en función del estado inicial, el algoritmo puede llegar a una solución u otra.
def plot_clusterer_comparison(clusterer1, clusterer2, X, title1=None, title2=None):
clusterer1.fit(X)
clusterer2.fit(X)
plt.figure(figsize=(10, 3.2))
plt.subplot(121)
plot_decision_boundaries(clusterer1, X)
if title1:
plt.title(title1, fontsize=14)
plt.subplot(122)
plot_decision_boundaries(clusterer2, X, show_ylabels=False)
if title2:
plt.title(title2, fontsize=14)
kmeans_rnd_init1 = KMeans(n_clusters=5, init="random", n_init=1,
algorithm="full", random_state=11)
kmeans_rnd_init2 = KMeans(n_clusters=5, init="random", n_init=1,
algorithm="full", random_state=19)
plot_clusterer_comparison(kmeans_rnd_init1, kmeans_rnd_init2, X,
"Solution 1", "Solution 2 (with a different random init)")
plt.show()
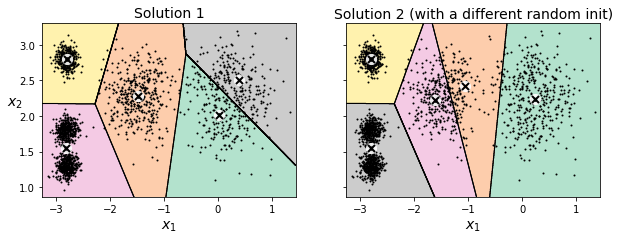
Para minimizar el impacto de la inicialización tenemos varias alternativas: probar varias y quedarnos con la mejor, o usar la inicialización inteligente que ofrece Scikit-Learn por defecto.
kmeans_rnd_10_inits = KMeans(n_clusters=5, init="random", n_init=10,
algorithm="full", random_state=11)
kmeans_rnd_10_inits.fit(X)
KMeans(algorithm='full', init='random', n_clusters=5, random_state=11)
plt.figure(figsize=(8, 4))
plot_decision_boundaries(kmeans_rnd_10_inits, X)
plt.show()
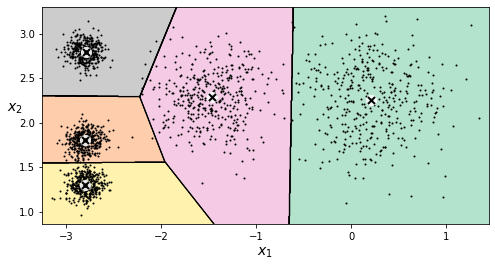
Otras mejoras al algoritmo de K-Means incluyen: K-Means acelerados (usado por Scikit-Learn por defecto y que evita algunos cálculos de distancias) y mini-batch K-Means (que puedes usarse si el dataset es tan grande que no cabe en memoria). Vamos a ver un ejemplo de este último caso con el dataset MNIST, para lo cual usaremos el objeto MiniBatchKMeans.
from sklearn.datasets import fetch_openml
mnist = fetch_openml('mnist_784', version=1)
mnist.target = mnist.target.astype(np.int64)
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(
mnist["data"], mnist["target"], random_state=42)
def load_next_batch(batch_size):
return X[np.random.choice(len(X), batch_size, replace=False)]
from sklearn.cluster import MiniBatchKMeans
k = 5
n_init = 10
n_iterations = 100
batch_size = 100
init_size = 500
evaluate_on_last_n_iters = 10
best_kmeans = None
for init in range(n_init):
minibatch_kmeans = MiniBatchKMeans(n_clusters=k, init_size=init_size)
X_init = load_next_batch(init_size)
minibatch_kmeans.partial_fit(X_init)
minibatch_kmeans.sum_inertia_ = 0
for iteration in range(n_iterations):
X_batch = load_next_batch(batch_size)
minibatch_kmeans.partial_fit(X_batch)
if iteration >= n_iterations - evaluate_on_last_n_iters:
minibatch_kmeans.sum_inertia_ += minibatch_kmeans.inertia_
if (best_kmeans is None or
minibatch_kmeans.sum_inertia_ < best_kmeans.sum_inertia_):
best_kmeans = minibatch_kmeans
best_kmeans.score(X)
-211.68949198767282
El algoritmo de mini-batch K-Means es más rápido que K-Means normal, pero su precisión suele ser peor.
Encontrando el número óptimo de grupos
Como hemos visto anteriormente, el número de grupos o clusters es un hyperparámetro del modelo que debemos definir, y como podrás imaginar si nuestros datos no están etiquetados no será imposible saber qué valor usar. Para ello la solución se basa en usar diferentes valores y utilizar alguna métrica significativa. Una de estas métricas se conoce como el silhouette score, que se basa en calcular el coeficiente de silueta (silhouette coefficient) de todas las muestras del dataset como donde es la distancia mínima al resto de muestras del mismo grupo y es la distancia media de los grupos más cercanos. Su valor se encuentra entre y . Un valor cercano a indica que la muestra está bien metida en su grupo y lejos de los demás mientras que un valor cercano a indica que la muestra se encuentra cerca de una frontera de decisión y un valor cercano a indica que la muestra está en el grupo incorrecto.
from sklearn.metrics import silhouette_score
silhouette_score(X, kmeans.labels_)
0.655517642572828
Calculando la métrica para diferentes números de clusters podemos encontrar el valor óptimo.
kmeans_per_k = [KMeans(n_clusters=k, random_state=42).fit(X)
for k in range(1, 10)]
silhouette_scores = [silhouette_score(X, model.labels_)
for model in kmeans_per_k[1:]]
plt.figure(figsize=(8, 3))
plt.plot(range(2, 10), silhouette_scores, "bo-")
plt.xlabel("$k$", fontsize=14)
plt.ylabel("Silhouette score", fontsize=14)
plt.axis([1.8, 8.5, 0.55, 0.7])
plt.show()
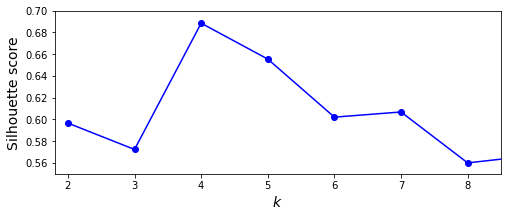
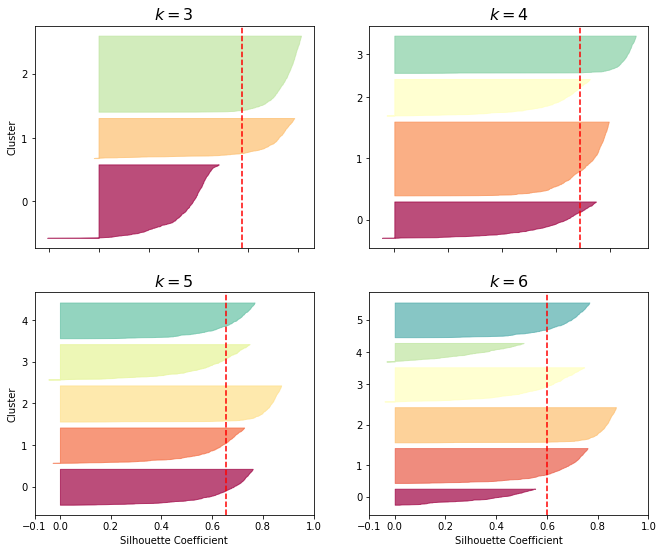
También es muy útil visualizar los diagramas de silueta, en el que visualizaremos todos los coeficientes de silueta ordenados por grupos y su valor.
from sklearn.metrics import silhouette_samples
from matplotlib.ticker import FixedLocator, FixedFormatter
import matplotlib as mpl
plt.figure(figsize=(11, 9))
for k in (3, 4, 5, 6):
plt.subplot(2, 2, k - 2)
y_pred = kmeans_per_k[k - 1].labels_
silhouette_coefficients = silhouette_samples(X, y_pred)
padding = len(X) // 30
pos = padding
ticks = []
for i in range(k):
coeffs = silhouette_coefficients[y_pred == i]
coeffs.sort()
color = mpl.cm.Spectral(i / k)
plt.fill_betweenx(np.arange(pos, pos + len(coeffs)), 0, coeffs,
facecolor=color, edgecolor=color, alpha=0.7)
ticks.append(pos + len(coeffs) // 2)
pos += len(coeffs) + padding
plt.gca().yaxis.set_major_locator(FixedLocator(ticks))
plt.gca().yaxis.set_major_formatter(FixedFormatter(range(k)))
if k in (3, 5):
plt.ylabel("Cluster")
if k in (5, 6):
plt.gca().set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
plt.xlabel("Silhouette Coefficient")
else:
plt.tick_params(labelbottom=False)
plt.axvline(x=silhouette_scores[k - 2], color="red", linestyle="--")
plt.title("$k={}$".format(k), fontsize=16)
plt.show()
K-Means para aprendizaje semi-supervisado.
El aprendizaje semi-supervisado (o Semi-supervised Learning) comprende el conjunto de técnicas que nos permiten entrenar modelos con datasets parcialmente etiquetados. En esta sección vamos a ver un ejemplo de como podemos aplicar esta técnica con el dataset MNIST y usando K-Means. Empezamos descargando el dataset.
from sklearn.datasets import fetch_openml
from sklearn.model_selection import train_test_split
import numpy as np
mnist = fetch_openml('mnist_784', version=1)
mnist.target = mnist.target.astype(np.int64)
X_train, X_test, y_train, y_test = train_test_split(
mnist["data"], mnist["target"], random_state=42)
Imagina que el dataset no estuviese etiquetado, es decir, que tuviésemos solo los dígitos sin la clase (sólo y no ) y tampoco supiésemos cuantas clases diferentes tenemos. Lo primero que podríamos hacer es entrenar un modelo de K-Means con, por ejemplos, 50 grupos. De esta manera encontraremos aquellas imágenes más representativas de cada grupo.
from sklearn.cluster import KMeans
k = 50
kmeans = KMeans(n_clusters=k, random_state=42)
X_digits_dist = kmeans.fit_transform(X_train)
idxs = np.argmin(X_digits_dist, axis=0)
X_representative_digits = X_train.values[idxs]
import matplotlib.pyplot as plt
plt.figure(figsize=(10, 5))
for index, X_representative_digit in enumerate(X_representative_digits):
plt.subplot(5, 10, index+1)
plt.imshow(X_representative_digit.reshape(28,28), cmap="binary")
plt.axis('off')
plt.show()

El siguiente paso consiste en anotar manualmente estas etiquetas (aquí haremos trampas ya que disponemos de dichas etiquetas :p).
y_representative_digits = y_train.values[idxs]
Y entrenaremos un clasificados usando estas imágenes representativas.
from sklearn.linear_model import LogisticRegression
log_reg2 = LogisticRegression(multi_class="ovr", solver="lbfgs", max_iter=5000, random_state=42)
%time log_reg2.fit(X_representative_digits, y_representative_digits)
log_reg2.score(X_test, y_test)
CPU times: user 844 ms, sys: 0 ns, total: 844 ms
Wall time: 87.7 ms
/home/juan/miniconda3/lib/python3.8/site-packages/sklearn/base.py:443: UserWarning: X has feature names, but LogisticRegression was fitted without feature names
warnings.warn(
0.7702285714285714
Como puedes observar hemos obtenido una precisión de casi el habiendo anotado simplemente 50 imágenes, pero aquellas más representativas. Entrenar el mismo modelo con 50 imágenes aleatorias nos daría peores resultados.
log_reg = LogisticRegression(multi_class="ovr", solver="lbfgs", max_iter=5000, random_state=42)
%time log_reg.fit(X_train[:50], y_train[:50])
log_reg.score(X_test, y_test)
CPU times: user 879 ms, sys: 15.4 ms, total: 895 ms
Wall time: 92.6 ms
0.5632571428571429
Esto pone de manifiesto que a la hora de entrenar modelos de ML no es tan importante la cantidad de datos, sino la calidad.
Ahora que tenemos un clasificador, podemos usarlo para anotar de manera automática el resto de imágenes. Para ello asignaremos, en cada grupo, la misma etiqueta a todas las muestras que la muestra representativa.
y_train_propagated = np.empty(len(X_train))
for i in range(k):
y_train_propagated[kmeans.labels_==i] = y_representative_digits[i]
log_reg3 = LogisticRegression(multi_class="ovr", solver="lbfgs", max_iter=5000, random_state=42)
%time log_reg3.fit(X_train[:1000], y_train_propagated[:1000])
log_reg3.score(X_test, y_test)
CPU times: user 10.4 s, sys: 164 ms, total: 10.6 s
Wall time: 1.08 s
0.7665142857142857
Esto sin embargo empeora nuestro clasificador, incluso usando más muestras de entrenamiento. Esto es debido al ruido que estamos introduciendo, ya que no todas las imágenes estarán bien agrupadas. Para mejorar esto podemos asignar etiquetas a aquellas muestras más cercanas a la imagen más representativa de cada grupo, o aplicar técnicas de aprendizaje activo.
Aprendizaje Activo
El aprendizaje activo (o Active Learning) consiste en entrenar modelos de ML de manera iterativa, incluyendo en cada iteración nuevas muestras al dataset focalizando en ejemplos en loa que el modelo tenga más problemas.
probas = log_reg3.predict_proba(X_train[:1000])
labels_ixs = np.argmax(probas, axis=1)
labels = np.array([proba[ix] for proba, ix in zip(probas, labels_ixs)])
sorted_ixs = np.argsort(labels)
labels[sorted_ixs[:10]]
array([0.99979563, 0.99982192, 0.99986221, 0.99986618, 0.99987415,
0.99987799, 0.99988065, 0.99989071, 0.99989191, 0.99989243])
X_lowest = X_train.values[:1000][sorted_ixs[:k]]
plt.figure(figsize=(10, 4))
for index, img in enumerate(X_lowest):
plt.subplot(k // 10, 10, index + 1)
plt.imshow(img.reshape(28, 28), cmap="binary", interpolation="bilinear")
plt.axis('off')
plt.show()

y_lowest = y_train.values[:1000][sorted_ixs[:k]]
y_lowest
array([7, 4, 9, 9, 4, 8, 4, 7, 3, 9, 5, 7, 7, 1, 3, 4, 4, 3, 4, 5, 6, 7,
2, 3, 2, 4, 0, 4, 8, 3, 4, 2, 4, 9, 5, 7, 9, 5, 2, 8, 5, 6, 2, 5,
4, 5, 3, 0, 0, 5])
y_train2 = y_train_propagated[:1000].copy()
y_train2[sorted_ixs[:k]] = y_lowest
log_reg5 = LogisticRegression(multi_class="ovr", solver="lbfgs", max_iter=5000, random_state=42)
%time log_reg5.fit(X_train[:1000], y_train2)
log_reg5.score(X_test, y_test)
CPU times: user 7.41 s, sys: 88.1 ms, total: 7.5 s
Wall time: 758 ms
0.7770285714285714
Podemos repetir el proceso tantas veces como haga falta hasta llegar a las prestaciones requeridas.
Otras técnicas de aprendizaje no supervisado
Otras técnicas muy usadas de aprendizaje no supervisado son
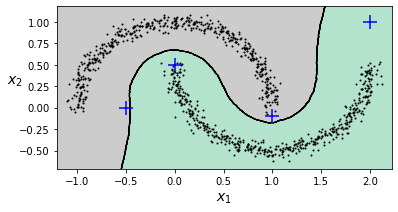
DBSCAN
Este algoritmo define clusters como regiones continuas de alta densidad, agrupando progresivamente muestras cercanas entre ellas.
from sklearn.datasets import make_moons
X, y = make_moons(n_samples=1000, noise=0.05, random_state=42)
from sklearn.cluster import DBSCAN
dbscan = DBSCAN(eps=0.05, min_samples=5)
dbscan.fit(X)
DBSCAN(eps=0.05)
np.unique(dbscan.labels_)
array([-1, 0, 1, 2, 3, 4, 5, 6])
dbscan2 = DBSCAN(eps=0.2)
dbscan2.fit(X)
np.unique(dbscan2.labels_)
array([0, 1])
def plot_dbscan(dbscan, X, size, show_xlabels=True, show_ylabels=True):
core_mask = np.zeros_like(dbscan.labels_, dtype=bool)
core_mask[dbscan.core_sample_indices_] = True
anomalies_mask = dbscan.labels_ == -1
non_core_mask = ~(core_mask | anomalies_mask)
cores = dbscan.components_
anomalies = X[anomalies_mask]
non_cores = X[non_core_mask]
plt.scatter(cores[:, 0], cores[:, 1],
c=dbscan.labels_[core_mask], marker='o', s=size, cmap="Paired")
plt.scatter(cores[:, 0], cores[:, 1], marker='*', s=20, c=dbscan.labels_[core_mask])
plt.scatter(anomalies[:, 0], anomalies[:, 1],
c="r", marker="x", s=100)
plt.scatter(non_cores[:, 0], non_cores[:, 1], c=dbscan.labels_[non_core_mask], marker=".")
if show_xlabels:
plt.xlabel("$x_1$", fontsize=14)
else:
plt.tick_params(labelbottom=False)
if show_ylabels:
plt.ylabel("$x_2$", fontsize=14, rotation=0)
else:
plt.tick_params(labelleft=False)
plt.title("eps={:.2f}, min_samples={}".format(dbscan.eps, dbscan.min_samples), fontsize=14)
plt.figure(figsize=(9, 3.2))
plt.subplot(121)
plot_dbscan(dbscan, X, size=100)
plt.subplot(122)
plot_dbscan(dbscan2, X, size=600, show_ylabels=False)
plt.show()

Podemos considerar las muestras fuera de los grupos como anomalías, o bien usar un clasificador de vecinos cercanos para clasificar nuevas muestras.
from sklearn.neighbors import KNeighborsClassifier
knn = KNeighborsClassifier(n_neighbors=50)
knn.fit(dbscan2.components_, dbscan2.labels_[dbscan2.core_sample_indices_])
KNeighborsClassifier(n_neighbors=50)
X_new = np.array([[-0.5, 0], [0, 0.5], [1, -0.1], [2, 1]])
knn.predict(X_new)
array([1, 0, 1, 0])
knn.predict_proba(X_new)
array([[0.18, 0.82],
[1. , 0. ],
[0.12, 0.88],
[1. , 0. ]])
plt.figure(figsize=(6, 3))
plot_decision_boundaries(knn, X, show_centroids=False)
plt.scatter(X_new[:, 0], X_new[:, 1], c="b", marker="+", s=200, zorder=10)
plt.show()
Gaussian Mixtures
Los modelos de Gaussian Mixtures son modelos probabilísticos que asumen que las muestras del conjunto de datos provienen de la mezcla de distribuciones Gaussianas cuyos parámetros son desconocidos.
X1, y1 = make_blobs(n_samples=1000, centers=((4, -4), (0, 0)), random_state=42)
X1 = X1.dot(np.array([[0.374, 0.95], [0.732, 0.598]]))
X2, y2 = make_blobs(n_samples=250, centers=1, random_state=42)
X2 = X2 + [6, -8]
X = np.r_[X1, X2]
y = np.r_[y1, y2]
plot_clusters(X)
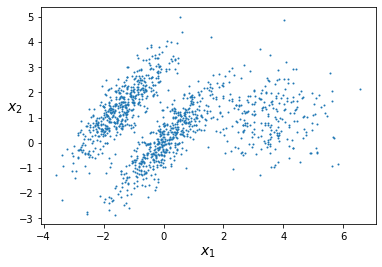
from sklearn.mixture import GaussianMixture
gm = GaussianMixture(n_components=3, n_init=10, random_state=42)
gm.fit(X)
GaussianMixture(n_components=3, n_init=10, random_state=42)
from matplotlib.colors import LogNorm
def plot_gaussian_mixture(clusterer, X, resolution=1000, show_ylabels=True):
mins = X.min(axis=0) - 0.1
maxs = X.max(axis=0) + 0.1
xx, yy = np.meshgrid(np.linspace(mins[0], maxs[0], resolution),
np.linspace(mins[1], maxs[1], resolution))
Z = -clusterer.score_samples(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z,
norm=LogNorm(vmin=1.0, vmax=30.0),
levels=np.logspace(0, 2, 12))
plt.contour(xx, yy, Z,
norm=LogNorm(vmin=1.0, vmax=30.0),
levels=np.logspace(0, 2, 12),
linewidths=1, colors='k')
Z = clusterer.predict(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.contour(xx, yy, Z,
linewidths=2, colors='r', linestyles='dashed')
plt.plot(X[:, 0], X[:, 1], 'k.', markersize=2)
plot_centroids(clusterer.means_, clusterer.weights_)
plt.xlabel("$x_1$", fontsize=14)
if show_ylabels:
plt.ylabel("$x_2$", fontsize=14, rotation=0)
else:
plt.tick_params(labelleft=False)
plt.figure(figsize=(8, 4))
plot_gaussian_mixture(gm, X)
plt.show()
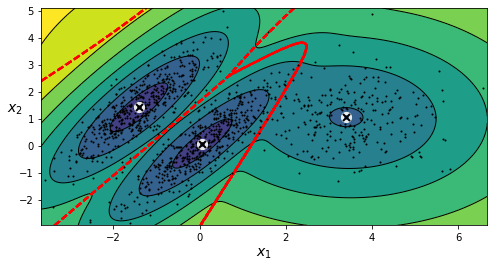
gm.weights_
array([0.39025715, 0.40007391, 0.20966893])
gm.means_
array([[ 0.05131611, 0.07521837],
[-1.40763156, 1.42708225],
[ 3.39893794, 1.05928897]])
gm.covariances_
array([[[ 0.68799922, 0.79606357],
[ 0.79606357, 1.21236106]],
[[ 0.63479409, 0.72970799],
[ 0.72970799, 1.1610351 ]],
[[ 1.14833585, -0.03256179],
[-0.03256179, 0.95490931]]])
gm.predict(X)
array([0, 0, 1, ..., 2, 2, 2])
gm.predict_proba(X)
array([[9.76741808e-01, 6.78581203e-07, 2.32575136e-02],
[9.82832955e-01, 6.76173663e-04, 1.64908714e-02],
[7.46494398e-05, 9.99923327e-01, 2.02398402e-06],
...,
[4.26050456e-07, 2.15512941e-26, 9.99999574e-01],
[5.04987704e-16, 1.48083217e-41, 1.00000000e+00],
[2.24602826e-15, 8.11457779e-41, 1.00000000e+00]])
Estos modelos son generativos, por lo que permiten crear nuevas muestras.
X_new, y_new = gm.sample(6)
X_new
array([[-0.86944074, -0.32767626],
[ 0.29836051, 0.28297011],
[-2.8014927 , -0.09047309],
[ 3.98203732, 1.49951491],
[ 3.81677148, 0.53095244],
[ 2.84104923, -0.73858639]])
y_new
array([0, 0, 1, 2, 2, 2])
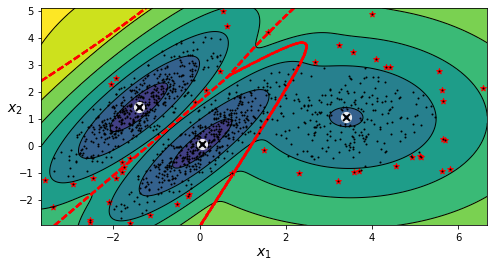
Definiendo un valor límite podemos detectar anomalías en los datos.
densities = gm.score_samples(X)
density_threshold = np.percentile(densities, 4)
anomalies = X[densities < density_threshold]
plt.figure(figsize=(8, 4))
plot_gaussian_mixture(gm, X)
plt.scatter(anomalies[:, 0], anomalies[:, 1], color='r', marker='*')
plt.ylim(top=5.1)
plt.show()
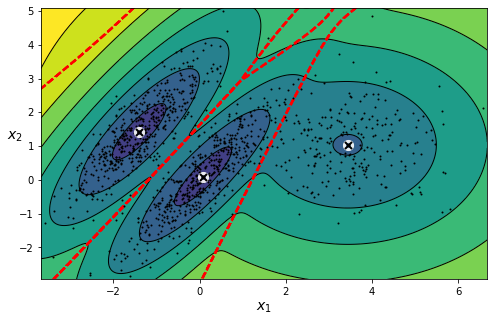
Una alternativa muy usada para seleccionar el número de grupos óptimos es el uso de Bayesian Gaussian Mixtures, ya que pueden asignar pesos cercanos a 0 a grupos innecesarios.
from sklearn.mixture import BayesianGaussianMixture
bgm = BayesianGaussianMixture(n_components=10, n_init=10, random_state=42)
bgm.fit(X)
BayesianGaussianMixture(n_components=10, n_init=10, random_state=42)
np.round(bgm.weights_, 2)
array([0.4 , 0.21, 0.4 , 0. , 0. , 0. , 0. , 0. , 0. , 0. ])
plt.figure(figsize=(8, 5))
plot_gaussian_mixture(bgm, X)
plt.show()
Resumen
En este post hemos introducido el concepto del aprendizaje no supervisado, modelos de Machine Learning que podemos usar cuando nuestros datos no están etiquetados. Hemos visto el algoritmo de K-Means y aplicado su uso en aprendizaje semi-supervisado y active learning. Por último, hemos introducido otros métodos útiles para detección de anomalías como DBSCAN o Gaussian Mixtures.